Systèmes d'adaptation des bactéries
Adaptation systems of bacteriaResearch
Multi-scale research: Molecular, cellular, community, applied science
Tools: microbiology, molecular biology, genetics, biochemistry, biophysics, omics
Bacterial models: Shewanella oneidensis, Escherichia coli, Staphylococcus aureus, Clostridium acetobutylicum, Nitratidesulfovibrio vulgaris, Pseudomonas aeruginosa, Neisseria sp…
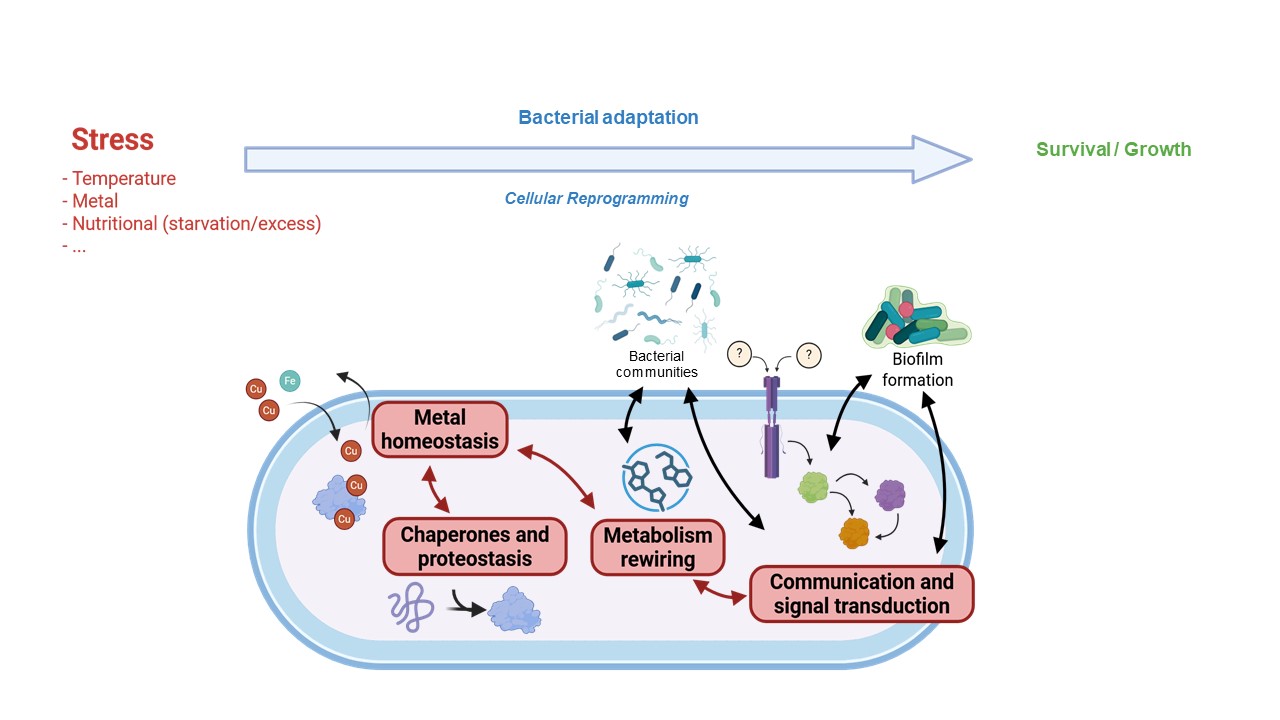
Les bactéries doivent s’adapter aux stress environnementaux qui perturbent le repliement et la stabilité des protéines. Pour faire face à ces défis, elles possèdent une classe conservée de protéines appelées chaperons, qui favorisent le repliement correct des protéines, empêchent leur agrégation, permettent leur désagrégation et régulent leur dégradation par les protéases.
Notre groupe s’intéresse aux chaperons (Hsp90, Hsp70/DnaK et ses co-chaperons de type JDP, Hsp33, etc.) dans divers modèles bactériens, notamment des espèces environnementales, commensales et pathogènes telles que Shewanella oneidensis, Escherichia coli et Pseudomonas aeruginosa. Nous cherchons à :
– Élucider les rôles des chaperons dans la physiologie bactérienne et l’adaptation au stress.
– Définir leurs réseaux d’interaction (clients, co-chaperons, autres chaperons, protéases).
– Découvrir leurs mécanismes d’action moléculaires, tant individuellement qu’en collaboration avec leurs partenaires.
Pour répondre à ces questions, nous utilisons un large éventail d’approches : biologie moléculaire, génétique, biochimie, -omique et biophysique.

Responsables scientifiques:
Sébastien DEMENTIN (Chargé de Recherche)
Olivier GENEST (Directeur de Recherche)
Marianne ILBERT (Directrice de Recherche)
Participants:
Elodie BERGE (Ingénieure d’Etude)
Safa BOUSSOUAR (Doctorante)
Ludovic DUBARD (Doctorant)
Alison FRATACCI (Doctorante)
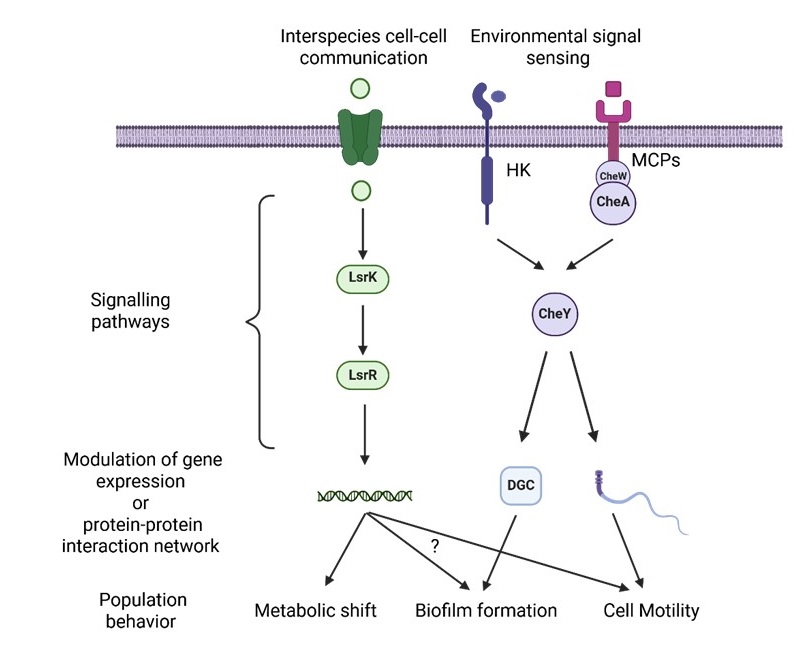
Les bactéries doivent adapter leur comportement en fonction des signaux perçus dans leur environnement. En réponse à ces signaux, elles peuvent se déplacer au sein de gradients de composés (chimiotactisme), former des structures communautaires complexes (biofilms) ou interagir avec d’autres espèces bactériennes (consortia). Pour ce faire, les bactéries possèdent des systèmes sophistiqués de détection et de transduction du signal.
Notre groupe s’intéresse aux voies de signalisation contrôlant, d’une part la biogenèse des biofilms et le chimiotactisme chez la bactérie environnementale Shewanella oneidensis, et d’autre part la communication intercellulaire au sein de consortia bactériens (E. coli, Clostridium acetobutylicum et Nitratidesulfovibrio vulgaris). Nous cherchons à :
– Identifier les acteurs des voies de signalisation.
– Définir les réseaux d’interaction au sein de ces voies.
– Comprendre les mécanismes moléculaires impliqués.
-Explorer les potentielles applications de biocontrôle des communautés microbiennes.
Pour répondre à ces questions, nous utilisons un large éventail d’approches : biologie moléculaire, génétique, biochimie, microscopie, -omique et biophysique.
Responsables scientifiques:
Cécile JOURLIN-CASTELLI (Maître de Conférences AMU)
Magali ROGER (Chargée de Recherche)
Participants:
Cassandra BACKES (Post-Doctorante)
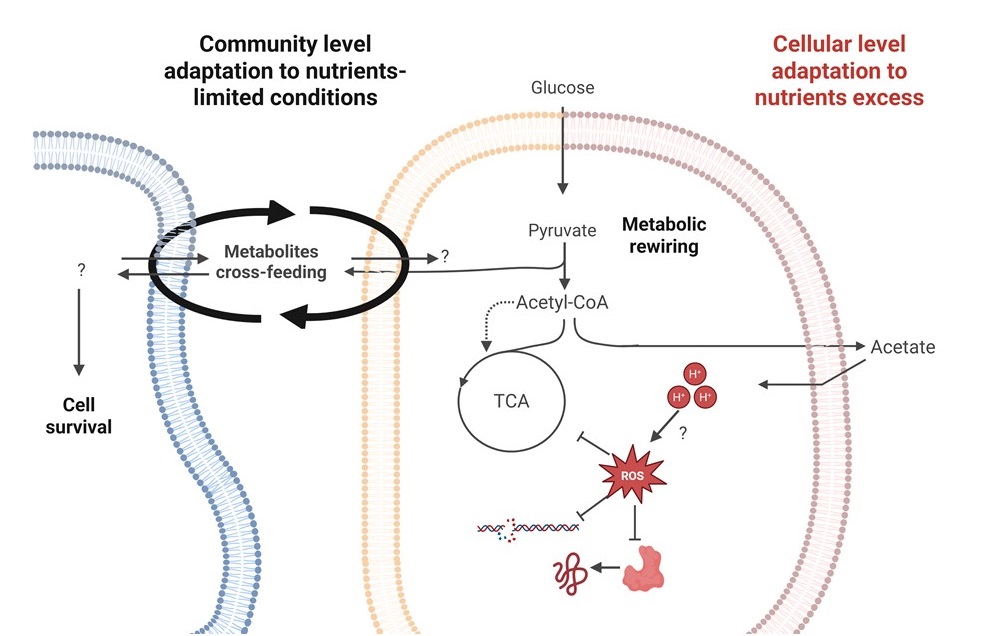
Les bactéries possèdent une remarquable capacité d’adaptation à une grande diversité de substrats, ce qui leur permet de coloniser un large éventail de niches écologiques, mais également de s’adapter aux variations auxquelles elles sont constamment exposées, qu’elles évoluent dans l’environnement ou au sein d’un hôte.
Lorsqu’elles subissent un stress nutritionnel, les bactéries sont capables de remodeler leur métabolisme afin d’optimiser l’utilisation des ressources disponibles, économiser l’énergie et favoriser leur survie. Ces réponses adaptatives reposent aussi bien sur des processus intracellulaires (reprogrammation transcriptionnelle et traductionnelle, réorientation des flux métaboliques) que des dynamiques collectives au sein de communautés bactériennes, où les interactions métaboliques inter-espèces contribuent à la résilience globale du système.
Nous abordons ces questionnements par une approche multi-échelle du gène à la communauté bactérienne afin d’obtenir une vision globale de la réponse adaptative associée à un stress nutritionnel chez différents modèles bactériens (S. aureus, E. coli, N. vulgaris, C. acetobutylicum).
-Responsables scientifiques:
David RANAVA (Maître de Conférences AMU)
Magali ROGER (Chargée de Recherche)
-Participants:
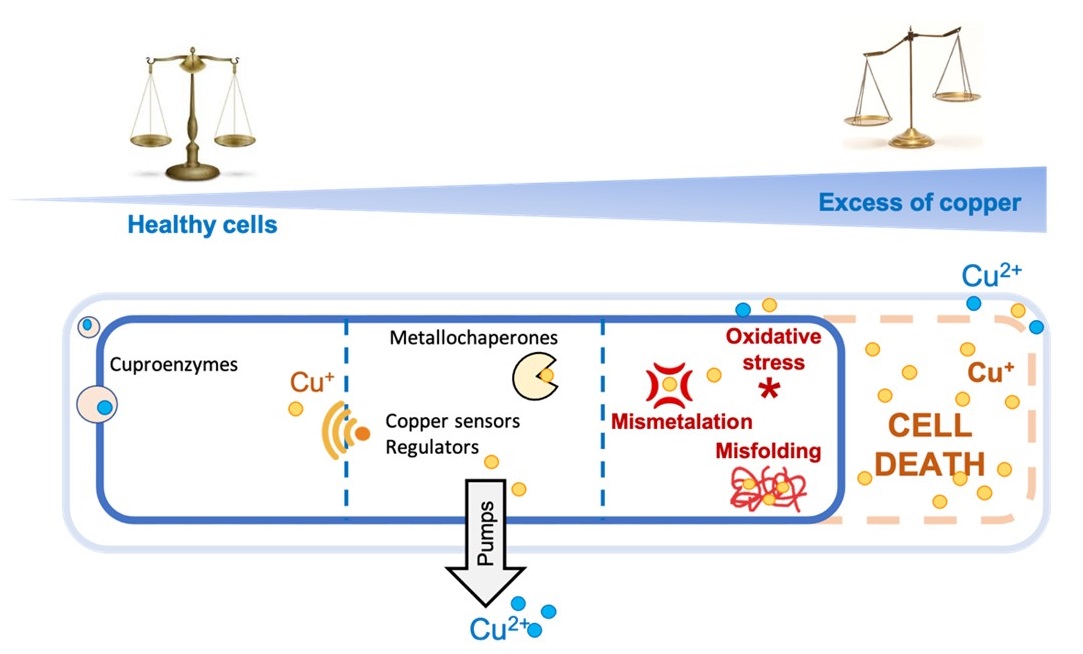
Les bactéries sont régulièrement confrontées à des variations de métaux tels que le cuivre, le zinc ou le fer. Bien que ces éléments soient indispensables comme cofacteurs enzymatiques, leur excès devient rapidement toxique en perturbant l’activité enzymatique, la structure des protéines et l’homéostasie cellulaire globale.
Notre équipe étudie comment les bactéries détectent, neutralisent et s’adaptent au stress métallique, avec un intérêt particulier pour le cuivre. Nos objectifs sont de :
- Définir les mécanismes moléculaires de la toxicité du cuivre, incluant la perturbation de la protéostasie et la mismétallation
- Caractériser les stratégies de régulation, de tolérance et de maintien de l’homéostasie du cuivre dans différents environnements
- Explorer le potentiel antimicrobien des complexes cuivre–ligand et élucider leurs modes d’action
Pour répondre à ces questions, nous combinons génétique bactérienne, microbiologie, biochimie, biophysique et des collaborations avec des chimistes, des structuralistes et des spécialistes de l’environnement.
Responsables scientifiques:
Marianne ILBERT (Directrice de Recherche)
Alex RIVERA-MILLOT (Chargé de Recherche)
Participants:















